데이터 전처리 (Data Preprocessing)
데이터 전처리는 데이터 사이언스, 머신 러닝 및 데이터 분석 프로젝트 필요한 데이터를 준비하고 가공하여 분석, 모델링 또는 머신 러닝 모델에 적합하게 사용할 수 있는 형태로 준비하는 포괄적인 과정
데이터의 품질을 향상시키고 분석 가능한 형태로 변환하여 모델의 성능을 최적화하고 정확성을 확보하기 위한 사전 단계
데이처 전처리 과정
1. 데이터 수집 : 프로젝트의 목적에 따라 적합한 데이터를 수집하고 데이터의 정확성과 완전성을 확인
- 시스템에서 데이터를 추출하더라도 엑셀 파일 등으로 변환하는 과정에서 누락이나 중복되는 경우 발생
- 데이터에 사용되는 여러 날짜 (예. 주문일, 물류센터 출고일, 고객 배송일 등)와 분석 대상 기간의 기준 확인 필요
2. 결측치 처리 : 데이터에 결측치(누락된 값)가 있는 경우, 이를 적절하게 처리
- 결측치 삭제 또는 대체
3. 이상치 처리 : 이상치(예외적인 값)를 탐지하고 이를 조치
4. 범주형 데이터 인코딩 : 범주형 변수를 수치형 변수로 변환
5. 데이터 분할 : 데이터를 학습용, 검증용 및 테스트용으로 구분
6. 데이터 정제 : 노이즈를 제거하거나 데이터에 오류를 수정
7. 데이터 표준화 : 데이터를 표준화하여 일관된 형식으로 변환
- 예) 날짜 형식을 통일하거나 단위를 표준화
데이터 분석의 80~90%는 전처리 작업, 나머지 10~20%는 전처리 작업 불평등하는데 쓴다는 말이 있을 정도로 분석 전에 하는 사전 작업에 많은 시간이 할애
궁극적으로 기계 학습 모델의 성능을 향상시켜 예측을 더 정확하게 수행하도록 하고, 모델 학습 및 평가에서 발생할 수 있는 문제를 사전에 식별하고 해결하므로 실질적인 시간과 자원을 절약하기 위함
데이터 클린징 (Data Cleansing) 프로세스
데이터 클린징은 데이터를 정확하고 유효하게 만드는 과정 (데이터 품질을 향상시켜 분석 결과의 신뢰성을 증대하는 과정)으로, 데이터 분석 전에 필수적인 과정
데이터에서 오류, 이상치 및 불필요한 정보(예: 잘못된 값, 중복 레코드)를 식별하고 수정 또는 제거하여 데이터의 정확성과 일관성을 향상
데이처 클린징 과정
1. 데이터 수집 : 데이터 클린징은 데이터 수집 단계에서 시작, 데이터 수집에 오류가 발생하지 않도록 주의
2. 결측치 처리 : 결측치(누락된 값)를 확인하고 대체하거나 제거
3. 이상치 처리 : 이상치(예외적인 값)를 탐지하고 수정하거나 제거
4. 중복 제거 : 중복된 레코드를 식별하고 제거
5. 데이터 형식 표준화 : 데이터 형식을 표준화하여 일관성을 유지
6. 데이터 유형 검사 : 데이터 유형(예: 숫자, 문자열)을 확인하고 잘못된 유형을 수정
7. 불필요한 열 제거 : 분석에 필요하지 않은 열을 제거
데이터 클린징(Data Cleansing)과 데이터 전처리(Data Preprocessing)는 유사한 작업을 포함하지만 중요한 차이점이 있음
- 데이터 전처리(Data Preprocessing) : 모델링에 적합한 형식으로 데이터 가공에 중점
- 데이터 클린징(Data Cleansing) : 데이터 품질을 개선하는 데 중점
결측치 (결측값, Missing Values) 처리 방법
해당 데이터를 삭제 또는 분석에서 제외
다른 값으로 대체
데이터 유형(성격)과 분석 목적에 따라 선택
1. 평균 또는 중앙값 대체 : 연속형 변수에 대한 일반적인 방법으로, 결측치를 해당 변수의 평균값 또는 중앙값으로 대체 (변수의 분포가 정규 분포와 유사한 경우에 효과적)
2. 최빈값 대체 : 범주형 변수에 대한 대체 방법으로, 결측치를 해당 변수의 최빈값으로 대체
3. 회귀 분석 기반 대체 : 다른 변수와의 관계를 고려하여 결측치를 예측하는 회귀 모델을 사용하여 대체
(다른 변수들과의 관계가 있는 경우 유용)
4. K-최근접 이웃(K-Nearest Neighbors) 대체 : 유사한 샘플을 찾아 해당 샘플들의 값으로 결측치를 대체
(데이터가 밀집된 경우에 유용)
5. 시계열 데이터의 경우 전방 또는 후방 채움 : 시간에 따라 변하는 데이터의 경우, 결측치를 해당 시점의 이전 값이나 이후 값으로 대체
6. 도메인 지식 기반 대체 : 해당 데이터와 관련된 도메인 지식을 활용하여 결측치를 대체
7. 다중 대체 방법 : 여러 대체 방법을 결합하여 사용하거나, 대체 후 성능을 비교하여 가장 적합한 대체 방법 선택
R 결측치 (결측값, Missing Values, NA) 처리
데이터에 NA가 포함되어 있으면, 결과값도 NA가 출력된다.
na.rm = TRUE 옵션을 사용하여 NA 값을 제외하고 요약 통계량을 계산할 수 있다.
특정 변수의 결측값을 확인하고 제거하기 위해서는 complete.cases함수 is.na 함수를 사용한다.
| install.packages('palmerpenguins') #팔머 펭귄 Dataset이 포함된 패키지 library(palmerpenguins) data("penguins") #Dataset 불러오기 summary(penguins) |
|||||||
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year |
| Adelie :152 | Biscoe :168 | Min. :32.10 | Min. :13.10 | Min. :172.0 | Min. :2700 | female:165 | Min. :2007 |
| Chinstrap: 68 | Dream :124 | 1st Qu.:39.23 | 1st Qu.:15.60 | 1st Qu.:190.0 | 1st Qu.:3550 | male :168 | 1st Qu.:2007 |
| Gentoo :124 | Torgersen: 52 | Median :44.45 | Median :17.30 | Median :197.0 | Median :4050 | NA's : 11 | Median :2008 |
| Mean :43.92 | Mean :17.15 | Mean :200.9 | Mean :4202 | Mean :2008 | |||
| 3rd Qu.:48.50 | 3rd Qu.:18.70 | 3rd Qu.:213.0 | 3rd Qu.:4750 | 3rd Qu.:2009 | |||
| Max. :59.60 | Max. :21.50 | Max. :231.0 | Max. :6300 | Max. :2009 | |||
| NA's :2 | NA's :2 | NA's :2 | NA's :2 | ||||
penguins 데이터는 총 344 Row로 구성되어 있으나, 일부 변수는 결측치가 포함됨
| penguins_df <- penguins no_col <- ncol(penguins_df) #변수별 결측값 수 확인 for(i in 1: no_col) { name_col <- names(penguins_df)[i] no_na <- sum ( is.na (penguins_df[,i]) ) cat ( name_col, ":", no_na, "\n") } |
| species : 0 island : 0 bill_length_mm : 2 bill_depth_mm : 2 flipper_length_mm : 2 body_mass_g : 2 sex : 11 year : 0 |
complete.cases(penguins_df)
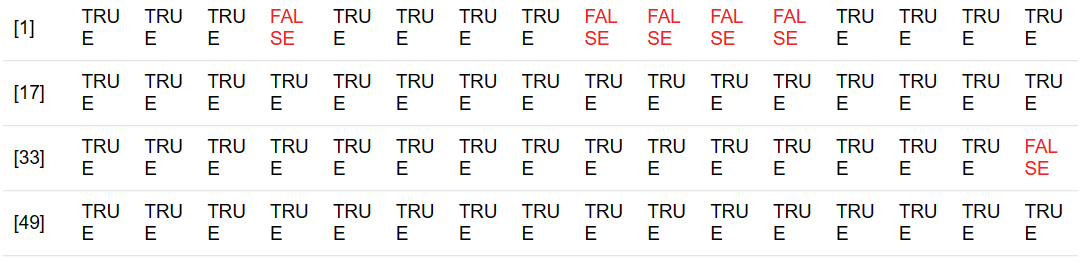
총 11개 행 (4, 9, 10, 11, 12, 48, 179, 219, 257, 269, 272 행)에 결측치 포함
결측치 제거 (complete.cases)
| penguins_df [!complete.cases(penguins_df),] | ||||||||
| # A tibble: 11 × 8 | ||||||||
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year | |
| <fct> | <fct> | <dbl> | <dbl> | <int> | <int> | <fct> | <int> | |
| 1 | Adelie | Torgersen | NA | NA | NA | NA | NA | 2007 |
| 2 | Adelie | Torgersen | 34.1 | 18.1 | 193 | 3475 | NA | 2007 |
| 3 | Adelie | Torgersen | 42 | 20.2 | 190 | 4250 | NA | 2007 |
| 4 | Adelie | Torgersen | 37.8 | 17.1 | 186 | 3300 | NA | 2007 |
| 5 | Adelie | Torgersen | 37.8 | 17.3 | 180 | 3700 | NA | 2007 |
| 6 | Adelie | Dream | 37.5 | 18.9 | 179 | 2975 | NA | 2007 |
| 7 | Gentoo | Biscoe | 44.5 | 14.3 | 216 | 4100 | NA | 2007 |
| 8 | Gentoo | Biscoe | 46.2 | 14.4 | 214 | 4650 | NA | 2008 |
| 9 | Gentoo | Biscoe | 47.3 | 13.8 | 216 | 4725 | NA | 2009 |
| 10 | Gentoo | Biscoe | 44.5 | 15.7 | 217 | 4875 | NA | 2009 |
| 11 | Gentoo | Biscoe | NA | NA | NA | NA | NA | 2009 |
| penguins_df2 <- penguins_df[complete.cases(penguins_df),] summary(penguins_df2) |
|||||||
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year |
| Adelie :146 | Biscoe :163 | Min. :32.10 | Min. :13.10 | Min. :172 | Min. :2700 | female:165 | Min. :2007 |
| Chinstrap: 68 | Dream :123 | 1st Qu.:39.50 | 1st Qu.:15.60 | 1st Qu.:190 | 1st Qu.:3550 | male :168 | 1st Qu.:2007 |
| Gentoo :119 | Torgersen: 47 | Median :44.50 | Median :17.30 | Median :197 | Median :4050 | Median :2008 | |
| Mean :43.99 | Mean :17.16 | Mean :201 | Mean :4207 | Mean :2008 | |||
| 3rd Qu.:48.60 | 3rd Qu.:18.70 | 3rd Qu.:213 | 3rd Qu.:4775 | 3rd Qu.:2009 | |||
| Max. :59.60 | Max. :21.50 | Max. :231 | Max. :6300 | Max. :2009 | |||
| str(penguins_df) str(penguins_df2) |
| tibble [344 × 8] (S3: tbl_df/tbl/data.frame) $ species : Factor w/ 3 levels "Adelie","Chinstrap",..: 1 1 1 1 1 1 1 1 1 1 ... $ island : Factor w/ 3 levels "Biscoe","Dream",..: 3 3 3 3 3 3 3 3 3 3 ... $ bill_length_mm : num [1:344] 39.1 39.5 40.3 NA 36.7 39.3 38.9 39.2 34.1 42 ... $ bill_depth_mm : num [1:344] 18.7 17.4 18 NA 19.3 20.6 17.8 19.6 18.1 20.2 ... $ flipper_length_mm: int [1:344] 181 186 195 NA 193 190 181 195 193 190 ... $ body_mass_g : int [1:344] 3750 3800 3250 NA 3450 3650 3625 4675 3475 4250 ... $ sex : Factor w/ 2 levels "female","male": 2 1 1 NA 1 2 1 2 NA NA ... $ year : int [1:344] 2007 2007 2007 2007 2007 2007 2007 2007 2007 2007 ... |
| tibble [333 × 8] (S3: tbl_df/tbl/data.frame) $ species : Factor w/ 3 levels "Adelie","Chinstrap",..: 1 1 1 1 1 1 1 1 1 1 ... $ island : Factor w/ 3 levels "Biscoe","Dream",..: 3 3 3 3 3 3 3 3 3 3 ... $ bill_length_mm : num [1:333] 39.1 39.5 40.3 36.7 39.3 38.9 39.2 41.1 38.6 34.6 ... $ bill_depth_mm : num [1:333] 18.7 17.4 18 19.3 20.6 17.8 19.6 17.6 21.2 21.1 ... $ flipper_length_mm: int [1:333] 181 186 195 193 190 181 195 182 191 198 ... $ body_mass_g : int [1:333] 3750 3800 3250 3450 3650 3625 4675 3200 3800 4400 ... $ sex : Factor w/ 2 levels "female","male": 2 1 1 1 2 1 2 1 2 2 ... $ year : int [1:333] 2007 2007 2007 2007 2007 2007 2007 2007 2007 2007 ... |
344 Row에서 333 Row로 감소
결측치 대체 (is.na, na.rm)
평균으로 대체
| penguins_df$bill_length_mm[is.na(penguins_df$bill_length_mm)] <- mean(penguins_df$bill_length_mm, na.rm = TRUE) penguins_df$bill_depth_mm[is.na(penguins_df$bill_depth_mm)] <- mean(penguins_df$bill_depth_mm, na.rm = TRUE) penguins_df$flipper_length_mm[is.na(penguins_df$flipper_length_mm)] <- mean(penguins_df$flipper_length_mm, na.rm = TRUE) |
|||||||
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year |
| Adelie :152 | Biscoe :168 | Min. :32.10 | Min. :13.10 | Min. :172.0 | Min. :2700 | female:165 | Min. :2007 |
| Chinstrap: 68 | Dream :124 | 1st Qu.:39.27 | 1st Qu.:15.60 | 1st Qu.:190.0 | 1st Qu.:3550 | male :168 | 1st Qu.:2007 |
| Gentoo :124 | Torgersen: 52 | Median :44.25 | Median :17.30 | Median :197.0 | Median :4050 | NA's : 11 | Median :2008 |
| Mean :43.92 | Mean :17.15 | Mean :200.9 | Mean :4202 | Mean :2008 | |||
| 3rd Qu.:48.50 | 3rd Qu.:18.70 | 3rd Qu.:213.0 | 3rd Qu.:4750 | 3rd Qu.:2009 | |||
| Max. :59.60 | Max. :21.50 | Max. :231.0 | Max. :6300 | Max. :2009 | |||
| NA's :2 | |||||||
상위 행 값으로 대체
| for (i in 2:length(penguins_df$sex)) { if (is.na(penguins_df$sex[i])) { penguins_df$sex[i] <- penguins_df$sex[i - 1] } } |
|||||||
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year |
| Adelie :152 | Biscoe :168 | Min. :32.10 | Min. :13.10 | Min. :172.0 | Min. :2700 | female:167 | Min. :2007 |
| Chinstrap: 68 | Dream :124 | 1st Qu.:39.27 | 1st Qu.:15.60 | 1st Qu.:190.0 | 1st Qu.:3550 | male :177 | 1st Qu.:2007 |
| Gentoo :124 | Torgersen: 52 | Median :44.25 | Median :17.30 | Median :197.0 | Median :4050 | Median :2008 | |
| Mean :43.92 | Mean :17.15 | Mean :200.9 | Mean :4202 | Mean :2008 | |||
| 3rd Qu.:48.50 | 3rd Qu.:18.70 | 3rd Qu.:213.0 | 3rd Qu.:4750 | 3rd Qu.:2009 | |||
| Max. :59.60 | Max. :21.50 | Max. :231.0 | Max. :6300 | Max. :2009 | |||
| NA's :2 | |||||||
다중 대체 방법 (sex에 따라, 평균값으로 대체)
| #암,수 평균 body_mass_g penguins_df %>% group_by (sex ) %>% summarise( mean_mass = mean(body_mass_g, na.rm = TRUE) ) -> penguins_df_mean # 결측치 대체 for (i in 1:nrow(penguins_df)) { if (is.na(penguins_df$body_mass_g[i])) { gender <- penguins_df$sex[i] if (gender == "male") { penguins_df$body_mass_g[i]<- penguins_df_mean$mean_mass[2] } else if (gender == "female") { penguins_df$body_mass_g[i] <- penguins_df_mean$mean_mass[1] } } } |
|||||||
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year |
| Adelie :152 | Biscoe :168 | Min. :32.10 | Min. :13.10 | Min. :172.0 | Min. :2700 | female:167 | Min. :2007 |
| Chinstrap: 68 | Dream :124 | 1st Qu.:39.27 | 1st Qu.:15.60 | 1st Qu.:190.0 | 1st Qu.:3550 | male :177 | 1st Qu.:2007 |
| Gentoo :124 | Torgersen: 52 | Median :44.25 | Median :17.30 | Median :197.0 | Median :4025 | Median :2008 | |
| Mean :43.92 | Mean :17.15 | Mean :200.9 | Mean :4200 | Mean :2008 | |||
| 3rd Qu.:48.50 | 3rd Qu.:18.70 | 3rd Qu.:213.0 | 3rd Qu.:4750 | 3rd Qu.:2009 | |||
| Max. :59.60 | Max. :21.50 | Max. :231.0 | Max. :6300 | Max. :2009 | |||
| > penguins_df_mean # A tibble: 2 × 2 sex mean_mass <fct> <dbl> 1 female 3862. 2 male 4518. |
| > penguins_df_mean$mean_mass[1] [1] 3862.273 |
| > penguins_df_mean$mean_mass[2] [1] 4518.22 |

'데이터 분석 (with Rstudio)' 카테고리의 다른 글
| [R Studio] 평균의 비교 T-검정(test) (논문 작성을 위한 네 번째 분석) (0) | 2023.09.28 |
|---|---|
| [R 기초] 기술통계 (논문 작성을 위한 세 번째 분석) (1) | 2023.09.26 |
| [R 기초] 교차 분석 (논문 작성을 위한 두 번째 분석) (0) | 2023.09.18 |
| [R 기초] 빈도 분석 (논문 작성을 위한 첫 번째 분석) (1) | 2023.09.18 |
| [R 기초] 변수 및 자료 유형 (벡터 및 주요함수) (0) | 2023.09.10 |



